|
|
il DNA: composizione,
funzioni e approfondimenti...
L'acido desossiribonucleico o deossiribonucleico (DNA)
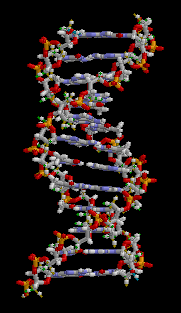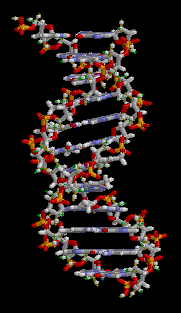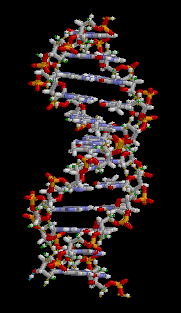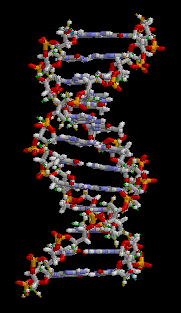
Il DNA è un lungo polimero costituito da unità ripetute di
nucleotidi. La catena del DNA è larga tra i 22 ed i 26 Ångström (da 2.2 a 2.6 nanometri) ed ogni unità nucleotidica è lunga 3.3 Ångstrom (0.33
nanometri). Sebbene ogni unità occupi uno spazio decisamente ridotto, la lunghezza dei polimeri di DNA può essere sorprendentemente elevata, dal momento che ogni filamento può contenere diversi milioni di nucleotidi. Ad esempio, il più grande cromosoma umano (il cromosoma 1) contiene quasi 250 milioni di paia di
basi.
Negli organismi viventi, il DNA non è quasi mai presente sotto forma di singolo filamento, ma come una coppia di filamenti saldamente associati tra
loro. Essi si intrecciano tra loro a formare una struttura definita doppia elica. Ogni nucleotide è costituito da uno scheletro laterale, che ne permette il legame covalente con i nucleotidi adiacenti, e da una base azotata, che instaura legami idrogeno con la corrispondente base azotata presente sul filamento opposto. Il composto formato da una base azotata legata allo zucchero è definito nucleoside; un nucleotide è invece un nucleoside a cui sono legati uno o più gruppi
fosfato.
La struttura laterale del DNA è composta da unità ripetute ed alternate di gruppi fosfato e di 2-deossiribosio,[24] uno zucchero pentoso (a cinque atomi di carbonio) che si lega ai fosfati adiacenti attraverso legami fosfodiesterici presso il terzo ed il quinto carbonio; in pratica, ogni molecola di fosfato forma un ponte molecolare collegando, attraverso legami fosfodiesterici, il carbonio in posizione 3' di una molecola di deossiribosio con quello in posizione 5' dello zucchero successivo. Conseguenza di questi legami asimmetrici è che ogni filamento di DNA ha un senso, determinato dalla direzione dei legami fosfodiesterici. In una doppia elica, il senso di un filamento è opposto a quello del filamento complementare. Per tale motivo, i due filamenti che costituiscono una doppia elica sono detti antiparalleli. Le estremità asimmetriche di un filamento di DNA sono definite estremità 5' (cinque primo) ed estremità 3' (tre primo). La principale differenza tra il DNA e l'RNA è lo zucchero pentoso utilizzato: l'RNA utilizza, infatti, il
ribosio.
La doppia elica del DNA è stabilizzata dai legami idrogeno che si instaurano tra le basi presenti sui due filamenti. Le quattro basi che sono state individuate nel DNA sono l'adenina (abbreviata con la lettera A), la citosina (C), la guanina (G) e la timina (T). Adenina e guanina sono composti eterociclici chiamati purine, mentre citosina e timina sono anelli
pirimidinici. Esiste una quinta base, di tipo pirimidinico, chiamata uracile (U), ma essa non è di norma presente nelle catene di DNA. L'uracile è altresì presente nei filamenti di RNA al posto della timina, da cui si differenzia per la mancanza di un gruppo metile. L'uracile è presente nel DNA solo come prodotto della degradazione della citosina. Solo nel batteriofago PBS1 tale base può essere utilizzata all'interno del
DNA. Al contrario, è molto più frequente individuare la timina all'interno di molecole di RNA, a causa della metilazione enzimatica di diversi uracili. Questo evento avviene solitamente a carico di RNA con funzione strutturale o enzimatica (rRNA e
tRNA).
La doppia elica è una spirale destrorsa. Con l'avvitarsi su sé stessi dei due filamenti, restano esposti dei solchi tra i diversi gruppi fosfato. Il solco maggiore è largo 22 Å, mentre il solco minore è largo 12 Å.[27] La differente ampiezza dei due solchi si traduce concretamente in una differente accessibilità delle basi, a seconda che si trovino nel solco maggiore o minore. Proteine come i fattori di trascrizione, dunque, solitamente prendono contatto con le basi presenti nel solco
maggiore.[
Approfondimento sull'appaiamento delle basi:
Ogni tipo di base presente su un filamento forma un legame con la base posta sul filamento opposto. Tale evento è noto come appaiamento complementare. Le basi puriniche formano legami idrogeno con le basi pirimidiniche: A può legare solo T e G può legare solo C. L'associazione di due basi viene comunemente chiamata paio di basi ed è l'unità di misura maggiormente utilizzata per definire la lunghezza di una molecola di DNA. Dal momento che i legami idrogeno non sono covalenti, essi possono esser rotti e riuniti in modo relativamente semplice. I due filamenti possono essere allontanati tra loro, come avviene per una cerniera, sia dalle alte temperature che da un'azione meccanica (come avviene durante la replicazione del
DNA). Conseguenza di questa complementarietà è che tutte le informazioni contenute nella doppia elica possono essere duplicate a partire da entrambi i filamenti, evento fondamentale per una corretta replicazione del
DNA.
I due tipi di paia di basi formano un numero differente di legami idrogeno: A e T ne formano due, G e C tre. Per tale motivo, la stabilità del legame GC è decisamente maggiore di quello AT. Di conseguenza, la stabilità complessiva di una molecola di DNA è direttamente correlata alla frequenza di GC presenti nella molecola stessa, nonché alla lunghezza dell'elica: una molecola di DNA è dunque tanto più stabile quanto più contiene GC ed è
lunga. Un'altra conseguenza di tale evento è il fatto che le regioni di DNA che devono essere separate facilmente contengono un'elevata concentrazione di A e T, come avviene ad esempio per il Pribnow box dei promotori batterici, la cui sequenza è infatti
TATAAT.
In laboratorio, la stabilità dell'interazione tra filamenti è misurata attraverso la temperatura necessaria a rompere tutti i legami idrogeno, chiamata temperatura di melting (o Tm). Quando tutti i legami idrogeno sono rotti, i singoli filamenti si separano e possono assumere strutture molto
variegate.
La stabilizzazione della doppia elica, in ogni caso, non è dovuta ai soli legami idrogeno, ma anche ad interazioni idrofobiche e di pi
stacking.
Cosa è il senso del DNA?
Una sequenza di DNA è definita senso se la sua sequenza è la stessa del relativo mRNA. La sequenza posta sul filamento opposto è invece detta antisenso. Dal momento che le RNA polimerasi lavorano producendo una copia complementare, il filamento necessario per la trascrizione è l'antisenso. Sia nei procarioti che negli eucarioti vengono prodotte numerose molecole di RNA antisenso a partire dalle sequenze senso. La funzione di questi RNA non codificanti non è stata ancora completamente
chiarita. Si ritiene che gli RNA antisenso possano giocare un ruolo nella regolazione dell'espressione
genica.
Esistono alcune sequenze di DNA, sia in procarioti che in eucarioti (ma soprattutto nei plasmidi e nei virus) in cui la differenza tra sequenze senso ed antisenso è meno chiara, dal momento che le sequenze di alcuni geni si sovrappongono tra
loro. In questi casi, dunque, alcune sequenze rivestono un doppio compito: codificare una proteina se lette in direzione 5'?3' su un filamento; codificarne un'altra se lette sull'altro (sempre in direzione 5'?3'). Nei batteri, questa sovrapposizione genica è spesso coinvolta nella regolazione della
trascrizione, mentre nei virus il fenomeno è dovuto alla necessità di contenere in un piccolo genoma un'elevata quantità di
informazioni. Un altro modo di ridurre le dimensioni genomiche è quello individuato da altri virus, che contengono molecole di DNA lineare o circolare a singolo filamento.
Quali sono i danni al DNA?
Il DNA può essere danneggiato dall'azione di numerosi agenti, genericamente definiti mutageni. Tra di essi figurano ad esempio agenti ossidanti, agenti alchilanti ed anche radiazioni ad alta energia, come i raggi X e gli UV. Il tipo di danno causato al DNA dipende dal tipo di agente: gli UV, ad esempio, danneggiano il DNA generando la formazione di dimeri di timina, costituiti da ponti aberranti che si instaurano tra basi pirimidiniche
adiacenti. Agenti ossidanti come i radicali liberi o il perossido di idrogeno, invece, producono danni di tipo più eterogeneo, come modificazioni di basi (in particolare di guanine) o rotture del DNA a doppio
filamento. Secondo diversi studi, in ogni cellula umana almeno 500 basi al giorno sono sottoposte a danni
ossidativi. Di tali lesioni, le più pericolose sono le rotture a doppio filamento, dal momento che tali danni sono i più difficili da riparare e costituiscono l'origine primaria delle mutazioni puntiformi e frameshift che si accumulano sulle sequenze genomiche, nonché delle traslocazioni
cromosomiche. Molti agenti devono il loro potere mutageno alla capacità di intercalarsi tra due basi azotate consecutive. Gli intercalanti sono tipicamente molecole planari e aromatiche, come l'etidio, la daunomicina, la doxorubicina o la talidomide. Perché un intercalante possa trovare posto tra le due basi, occorre che la doppia elica si apra e perda la sua conformazione standard. Tali modifiche strutturali inibiscono sia la trascrizione che la replicazione del DNA ed aumentano la possibilità di insorgenza di mutazioni. Per tale motivo, gli intercalanti sono considerati molecole cancerogene, come dimostrato da numerosi studi su molecole come il benzopirene, l'acridina, l'aflatossina ed il bromuro di
etidio. In ogni caso, proprio grazie alla loro capacità di inibire trascrizione e replicazione, tali molecole sono anche utilizzate in chemioterapia per inibire le cellule neoplastiche a rapida crescita.
Quali sono le funzioni del DNA?
Negli eucarioti, il DNA è solitamente presente all'interno di cromosomi lineari (circolari nei procarioti). La somma di tutti i cromosomi di una cellula ne costituisce il genoma; il genoma umano conta circa 3 miliardi di paia di basi contenute in 46
cromosomi. L'informazione è conservata in sequenze di DNA chiamate geni. La trasmissione dell'informazione contenuta nei geni è garantita dalla presenza di sequenze di basi azotate complementari. Infatti, durante la trascrizione, l'informazione può essere facilmente copiata in un filamento complementare di RNA. Solitamente, tale copia di RNA è utilizzata per sintetizzare una proteina, attraverso un processo definito traduzione (o sintesi proteica). In alternativa, una cellula può semplicemente duplicare l'informazione genetica attraverso un processo definito replicazione del DNA.
Definizione delle struttura del genoma:
Negli organismi eucarioti, il DNA genomico è localizzato all'interno del nucleo cellulare, nonché in piccole quantità all'interno di mitocondri e cloroplasti. Nei procarioti, il DNA è invece racchiuso in un organello irregolare, privo di membrana, contenuto nel citoplasma, chiamato
nucleoide. L'informazione è contenuta all'interno dei geni, unità ereditarie in grado di influire sul fenotipo dell'organismo. Ogni gene contiene un open reading frame (regione in grado di essere trascritta a RNA) ed una regione regolatoria, costituita sia da un promotore che da
enhancers.
In molte specie, solo una piccola frazione della sequenza totale di un genoma può essere trascritta e tradotta. Ad esempio, solo l'1.5% del genoma umano è costituito da esoni codificanti per una proteina, mentre più del 50% consiste di sequenze ripetute di DNA non
codificante. La ragione per cui ci sia una tale quantità di DNA non codificante non è tuttora completamente chiara ed è stata definita come enigma del
C-value. In ogni caso, le sequenze di DNA che non codificano per una proteina possono essere trascritte in RNA non codificante, coinvolto nella regolazione dell'espressione
genica.[
Alcune sequenze non codificanti ricoprono un ruolo strutturale per i cromosomi. Le regioni telomeriche e centromeriche contengono solitamente pochissimi geni, ma sono necessarie per la funzione e la stabilità dei
cromosomi. Nell'uomo, grandi quantità di DNA non codificante si ritrovano negli pseudogeni, copie di geni rese inattive dalla presenza di una
mutazione. Queste sequenze sono considerate come fossili molecolari, anche se esistono evidenze secondo le quali si può ipotizzare che siano una sorta di materiale grezzo necessario per la creazione di nuovi geni attraverso i processi di duplicazione genica e di evoluzione
divergente.
La trasduzione e la traduzione del DNA:
Un gene è una sequenza di DNA che contiene le informazioni in grado di influire sulle caratteristiche del fenotipo dell'organismo. All'interno di un gene, la sequenza di basi di DNA è utilizzata come stampo per la sintesi di una molecola di RNA che, nella maggior parte dei casi, è tradotta in una molecola
peptidica. Il meccanismo attraverso il quale la sequenza nucleotidica di un gene è copiata in un filamento di RNA è detto trascrizione ed avviene per mezzo dell'enzima RNA polimerasi. Il filamento di RNA può andare incontro a destini differenti: alcune molecole di RNA hanno, infatti, funzioni di tipo strutturale (come quelle che si trovano all'interno del ribosoma) o catalitica (molecole note come ribozimi); la maggior parte degli RNA, tuttavia, subiscono un processo di maturazione per produrre mRNA, molecole destinate ad esser tradotte in proteina.
Il processo di traduzione avviene nel citoplasma, dove gli mRNA si associano ai ribosomi, ed è mediato dal codice genetico. Il ribosoma permette la lettura sequenziale dei codoni del mRNA, favorendone il riconoscimento e l'interazione con specifici tRNA, molecole che trasportano gli amminoacidi corrispondenti ad ogni singolo
codone.
Approfondimento sul codice genetico:
Complementarietà delle basi azotate del DNA vista in dettaglio.Il codice genetico consiste di parole di tre lettere chiamate codoni, costituite dalla sequenza di tre nucleotidi (ad esempio ACT, CAG, TTT), ognuna delle quali è associata ad un particolare amminoacido. Ad esempio la timina ripetuta in una serie di tre (TTT) codifica per la fenilalanina. Utilizzando gruppi di tre lettere si possono avere fino a 64 combinazioni diverse (43), in grado di codificare per i venti diversi amminoacidi esistenti. Poiché esistono 64 triplette possibili e solo 20 amminoacidi, il codice genetico è detto ridondante (o degenere): alcuni amminoacidi possono infatti essere codificati da più triplette diverse. Non è invece vero il contrario: ad ogni tripletta corrisponderà un solo amminoacido (senza possibilità di ambiguità). Esistono infine tre triplette che non codificano per nessun amminoacido, ma rappresentano codoni di stop (o nonsense), ovvero indicano il punto in cui, all'interno del gene, termina la sequenza che codifica per la proteina corrispondente: si tratta dei codoni TAA, TGA e
TAG.
Quali sono gli anzimi che possono modificare il
DNA?
Nucleasi e Ligasi
Le nucleasi sono enzimi in grado di tagliare filamenti di DNA, dal momento che catalizzano l'idrolisi del legame fosfodiesterico. Le nucleasi che idrolizzano il DNA partendo dai nucleotidi situati alle estremità dei filamenti sono definite esonucleasi. Sono endonucleasi, invece, quelle che tagliano direttamente all'interno del filamento. Le nucleasi più utilizzate in biologia molecolare, dette enzimi di restrizione, tagliano il DNA in corrispondenza di specifiche sequenze. L'enzima EcoRV, ad esempio, riconosce la sequenza di sei basi 5'-GAT|ATC-3' ed effettua il taglio presso la linea verticale. In natura, questo enzima protegge i batteri dalle infezioni fagiche, digerendo il DNA del fago quando esso fa il suo ingresso nella cellula
batterica. Generalmente, le nucleasi di restrizione riconoscono particolari sequenze nucleotidiche palindromiche, note come siti di restrizione, nelle quali la stessa sequenza si ripete in direzioni opposte sulle due eliche complementari, e quindi producono dei tagli su entrambi i filamenti. Tali enzimi sono utilizzati ampiamente nelle tecniche che prevedono il subclonaggio di DNA all'interno di vettori.
Le DNA ligasi sono enzimi in grado di riunire filamenti di DNA precedentemente tagliati o spezzati, utilizzando energia chimica proveniente da ATP o da
NAD. Le ligasi sono particolarmente importanti nella replicazione del filamento lento, dal momento che esse riuniscono i frammenti di Okazaki in un filamento unico. Esse rivestono un ruolo importante anche nella riparazione del DNA e nella ricombinazione
genetica.
Topoisomerasi ed Elicasi
Le topoisomerasi sono enzimi che presentano sia un'attività nucleasica che una ligasica. Queste proteine sono in grado di modificare le proprietà topologiche del DNA. Alcune di esse svolgono tale funzione tagliando l'elica di DNA e permettendole di ruotare, riducendo il suo grado di superavvolgimento, per poi procedere alla ligazione delle due estremità. Altre topoisomerasi sono invece in grado di tagliare l'elica e far passare attraverso il sito di rottura una seconda elica, prima di ligare il filamento
spezzato. Le topoisomerasi sono necessarie per molti processi che coinvolgono il DNA, come ad esempio la replicazione del DNA e la
trascrizione.
Le elicasi sono proteine in grado di utilizzare l'energia chimica presente nei nucleosidi trifosfato, soprattutto ATP, per rompere i legami idrogeno che si instaurano tra le basi azotate, permettendo l'apertura della doppia elica di DNA in singoli
filamenti. Questi enzimi sono essenziali per la maggior parte dei processi biologici che coivolgono enzimi che richiedono un diretto contatto con le basi del DNA.
Polimerasi
Le polimerasi sono enzimi che sintetizzano catene polinucleotidiche a partire dal nucleosidi trifosfato. Esse funzionano aggiungendo nucleotidi al 3'-OH del precedente nucleotide presente sul filamento. Come conseguenza di ciò, tutte le polimerasi lavorano in direzione
5'->3'. Nel sito attivo di questi enzimi, il nucleoside trifosfato si appaia ad un nucleotide presente su un filamento usato come stampo: ciò permette alle polimerasi di sintetizzare in modo accurato filamenti fedelmente complementari agli stampi. Le polimerasi sono classificate sulla base del tipo di stampo che utilizzano.
La replicazione del DNA richiede una DNA polimerasi DNA-dipendente, in grado cioè di realizzare una perfetta copia di una sequenza di DNA. L'accuratezza è fondamentale in questo processo, motivo per cui molte di queste polimerasi presentano anche un'attività di proofreading (dall'inglese, correzione di bozze). Esse sono infatti in grado di rilevare un errore di appaiamento (o mismatch) tra basi azotate ed attivare un'azione 3' o 5' esonucleasica per rimuovere la base
scorretta. Nella maggior parte degli organismi, le DNA polimerasi funzionano all'interno di un più ampio complesso proteico definito replisoma, che consiste anche di numerose subunità accessorie come ad esempio le
elicasi.
Le DNA polimerasi RNA-dipendenti sono una classe di polimerasi specializzate nella sintesi di una copia di DNA, usando come stampo un frammento di RNA. Tra di esse figurano la trascrittasi inversa, un enzima virale coinvolto nell'infezione dei retrovirus, e la telomerasi, necessaria per la replicazione dei
telomeri. La telomerasi è una polimerasi inusuale, dal momento che contiene una sequenza stampo di RNA all'interno della propria struttura.
La trascrizione è invece svolta da RNA polimerasi DNA-dipendenti, che legano il DNA presso il promotore di un gene e separano i due filamenti. Successivamente, l'enzima genera una molecola di mRNA fino al raggiungimento del terminatore, dove si interrompe la trascrizione e l'enzima si distacca dal DNA. Come avviene per le DNA polimerasi DNA-dipendenti, anche queste polimerasi operano all'interno di un ampio complesso proteico, composto di molecole accessorie e
regolatorie.
Approfondimento sull'evoluzione del metabolismo del
DNA:
Il DNA contiene l'informazione genetica che permette a tutti gli organismi viventi (esclusi i virus, la cui ammissibilità tra i viventi è tuttavia ampiamente dibattuta) di funzionare, crescere e riprodursi. In ogni caso, non è stato ancora chiarito in quale momento della storia della vita il DNA abbia assunto tale ruolo fondamentale. È generalmente accettato dalla comunità scientifica, infatti, l'ipotesi che il DNA non sia stato il primo acido nucleico ad essere utilizzato dai viventi: tale ruolo spetterebbe infatti
all'RNA. L'RNA potrebbe aver giocato un ruolo centrale del metabolismo cellulare ancestrale, dal momento che può avere sia un ruolo nella conservazione dell'informazione genetica, sia uno catalitico (ad esempio attraverso i ribozimi), sia uno strutturale (all'interno dei
ribosomi). Il mondo a RNA, basato su un unico tipo di molecola avente funzioni genetiche, catalitiche e strutturali, potrebbe avere avuto un'influenza sullo sviluppo dell'attuale codice genetico, basato proprio su quattro nucleotidi. Tale numero potrebbe essere un compromesso tra la necessità da una parte di ridurre la quantità di basi possibili, per migliorare l'accuratezza della replicazione, e dall'altra di aumentarla, per incrementare l'efficacia catalitica dei
ribozimi. In ogni caso, non esistono prove evidenti di questo sistema genetico ancestrale, dal momento che è impossibile recuperare DNA dai fossili. Ciò è dovuto al fatto che il DNA può sopravvivere nell'ambiente per meno di un milione di anni e, se in soluzione, si degrada rapidamente in piccoli
frammenti. Sebbene ci siano stati diversi annunci di scoperte di DNA antichissimo, tra cui quella relativa all'isolamento di un batterio vivo da un cristallo di sale risalente a 250 milioni di anni
fa, queste affermazioni sono ancora controverse e oggetto di discussione.
Sito web consigliato:
WIKIPEDIA,
l'enciclopedia libera...

Questo articolo è protetto dalle Leggi Internazionali di Proprietà.
E' PROIBITA la sua riproduzione totale o parziale, all'interno di qualsiasi mezzo di comunicazione
(cartaceo, elettronico, ecc.) senza l'autorizzazione scritta dell'autore.
|
